Metagenômica: o “projeto genoma” dos micróbios
Nos últimos anos, a Biologia viu surgir uma infinidade de novos campos e subcampos de pesquisa inspirados na genômica, as “ômicas”. O que as ômicas têm em comum é a quantificação e caracterização de determinadas moléculas dos seres vivos, determinando “perfis” ou padrões dessas moléculas entre os organismos e buscando causas para as diferenças encontradas. Entre as ômicas, há a genômica (que lida com moléculas de DNA), a transcriptômica (com RNAs, ou transcriptos), a proteômica (enzimas e outras proteínas) e a metabolômica (metabólitos, como açúcares e lipídios, dentre outros).
Destas, a mais famosa, sem dúvida, é a genômica, devido ao projeto Genoma Humano, realizado em conjunto por várias instituições de pesquisa. O projeto, iniciado em 1990, decifrou a sequência de todo o DNA humano, necessitando de 13 anos para ficar pronto, do envolvimento de milhares de pessoas (dentre pesquisadores e técnicos), do investimento de 3,8 bilhões de Dólares, e resultando numa ‘’enciclopédia’’ que ocupa uma estante toda!
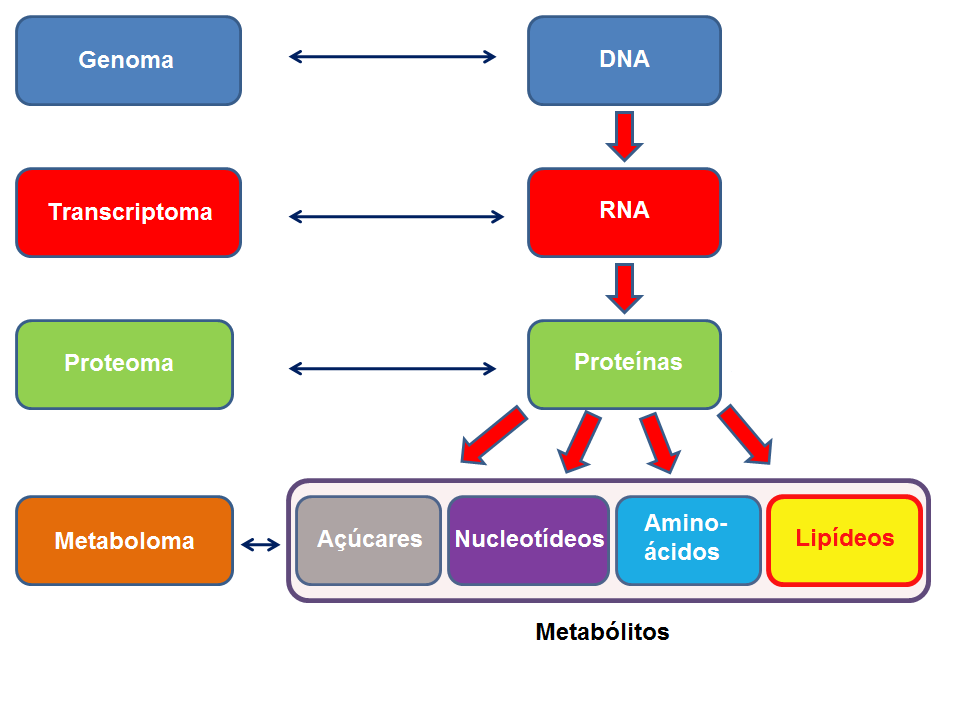 |
| Ômicas – Para cada conjunto de biomoléculas, surgiu um novo campo de pesquisa entre as ômicas. Antes das ômicas, esses compostos já eram estudados em disciplinas como a biologia molecular (os DNAs, RNAs e proteínas) e a bioquímica (as proteínas e os outros metabólitos), porém, de forma mais individualizada, e não em conjunto. Modificado de: http://en.wikipedia.org/wiki/File:Metabolomics_schema.png (CC BY-SA 3.0). |
Sequenciar um genoma (o DNA total de um organismo) é algo muito custoso e demorado, além de ser complicado lidar com toda a informação gerada – ainda que não passem de sequências de letras A, T, C e G, que representam os nucleotídeos, ou “tijolos”, de um DNA. No entanto, novas técnicas de sequenciamento e o aprimoramento do campo da bioinformática vêm mudando isso, tanto que tornaram possível o sequenciamento do genoma de vários outros organismos, além de que uma nova ômica se concretizasse: a metagenômica ou genômica ambiental.
Esse novo campo de pesquisas busca fazer simultaneamente o sequenciamento do genoma de muitos organismos (neste caso, microorganismos) obtidos de uma amostra ambiental – por exemplo, do solo, da água do mar, do ar, ou da sua própria pele ou boca, já que nós somos um viveiro de microorganismos. Se, em uma gota de água do mar, pode haver milhões de microorganismos, às vezes com genomas maiores que o humano, imagine as “bibliotecas” geradas nesses estudos – as quais, obviamente, não são impressas, mas ficam armazenadas em bancos de dados disponíveis na Internet, como o GenBank.
Entre as milhões de espécies de bactérias, protistas (algas e protozoários), e mesmo vírus, muitas sequências de DNA são iguais, mas outras são únicas de cada espécie, funcionando como marcadores que permitem a identificação, na “biblioteca” de genomas gerada, de quais organismos estavam presentes na amostra estudada. Pode-se então fazer comparações com amostras de outros ambientes, tentando-se entender porque as composições de espécies são diferentes. Um problema é que muitas espécies de microorganismos ainda não têm marcadores, porque ainda não foram estudadas para isto. Para esses estudos, é preciso isolar o microorganismo e fazer um sequenciamento individual do material genético, mas não necessariamente do DNA inteiro.
No entanto, nos estudos de metagenômica, nem sempre os pesquisadores estão preocupados em saber a identidade das espécies de microorganismos presentes na amostra. Muitas vezes, eles querem saber apenas quais sequências de DNA estão presentes, pois isso, por si só, basta para saber quais proteínas existem naquele ambiente. Isto permite inferir, por exemplo, se tal ambiente é mais produtor de matéria orgânica (como um pasto de microorganismos) ou consumidor (um restaurante de micróbios). No entanto, como ainda é um campo recente, a maioria dos estudos se ocupa mais de documentar os metagenomas de cada ambiente do que em tecer teorias a respeito dos padrões observados.
Humboldts do Século XXI
Assim, podemos comparar certos pesquisadores de metagenômica com os naturalistas – nome que se dava aos pesquisadores que eram uma mistura de biólogos e geólogos – que, há 200 anos, vinham aos trópicos para documentar nossa imensa riqueza de plantas e animais, que, em geral, são “macroorganismos”. Muitos desses naturalistas se ocupavam em descobrir novas espécies – o que se chama taxonomia, ou sistemática; outros queriam saber os locais onde cada organismo estava presente – biogeografia.
 |
| Cópia impressa do genoma humano, no museu Wellcome Collection, Londres. (Fonte: http://commons.wikimedia.org/wiki/File:Wellcome_genome_bookcase.png (CC BY-SA 3.0).) |
A “biogeografia macrobiana” é hoje um campo de pesquisas maduro. Sabe-se, por exemplo, que algumas espécies são cosmopolitas, como os pardais, e que a maioria tem distribuição limitada, como as jabuticabas na América do Sul ou as girafas na África. Já a “biogeografia microbiana” está só engatinhando, por dois motivos principais. Primeiro porque, até recentemente, para se estudar alguns microorganismos como as bactérias, a única maneira que havia era isolá-los de sua amostra e esperá-los crescerem num novo recipiente, formando colônias, e só então fazer testes para identificar quais espécies estavam presentes nas amostras (alguns outros microorganismos, como protistas, são em geral mais fáceis de identificar, bastando olhá-los ao microscópio). Só que, em isolamento, em geral, a maior parte (95%) das bactérias de uma amostra não se multiplicam, e assim, se não formam colônias, não há como identificá-las e nem saber sua biogeografia.
O outro motivo é que há uma teoria centenária, chamada hipótese de Baas-Becking, segundo a qual, entre os microorganismos, “tudo está em todo lugar, mas o ambiente seleciona”. Em outras palavras, os microorganismos seriam todos cosmopolitas, pois têm uma grande capacidade de reprodução e dispersão, e o único limitante à sua distribuição seriam as condições ambientais. Assim, uma espécie de ameba que ocorre em lagos limpos deveria ocorrer em todos os lagos do mundo, desde que sejam limpos. Ou ainda, um lago limpo deveria conter todas as espécies de microorganismos capazes de viver em lagos limpos existentes no mundo todo. Inicialmente, alguns estudos (mesmo com o problema do isolamento) confirmaram isto para alguns grupos de microorganismos. Só que isso foi generalizado para todos eles, e a questão de se os microorganismos possuem ou não distribuição limitada ficou um pouco esquecida por muito tempo, até que a metagenômica surgisse. Até o momento, estudos de metagenômica já revelaram que pelo menos alguns microorganismos possuem sim distribuição limitada e não são cosmopolitas. Este é um exemplo de que, nas Ciências da Natureza, não é apenas a técnica (ou a falta de) que determina os rumos das pesquisas, mas também as ideias compartilhadas pelos pesquisadores.
Pesquisa aplicada
Como já foi dito, tendo as sequências de DNA, é possível saber também quais as enzimas produzidas pelos microorganismos. As enzimas, um tipo de proteína, são moléculas produzidas nas células, com base nas sequências de DNA. Elas aceleram reações químicas específicas, como, por exemplo, a degradação de substâncias em nosso organismo. Muitas têm interesse para o ser humano, que é capaz de produzi-las em larga escala, através de engenharia genética (manipulação de genomas). Na engenharia genética, isola-se a sequência de DNA que produz a enzima de interesse e modifica-se a sequência para que a enzima seja produzida em maior quantidade do que o normal. Por fim, insere-se o DNA novamente em uma bactéria ou levedura (um fungo).
Alguns pesquisadores estão agora usando a metagenômica para encontrar enzimas mais eficientes. Uma cobiçada enzima é a celulase, que transforma a celulose em combustível. A celulose é o principal componente, por exemplo, do bagaço da cana, que é descartado durante a produção de álcool e açúcar. Já existem celulases produzidas por engenharia genética, mas ainda não são muito boas. Um local de grande procura por essas enzimas é o intestino de ruminantes, pois nele há muitas bactérias que produzem celulases.
Os microorganismos do solo de áreas de vegetação de cerrado têm também potencial de apresentar enzimas de interesse biotecnológico. Esse solo tem muitos metais pesados e tóxicos, e os microorganismos precisam de mecanismos para contornar isso. Dentre tais mecanismos, estão suas enzimas, que podem ser úteis em biorremediação – a despoluição de áreas degradadas usando organismos.
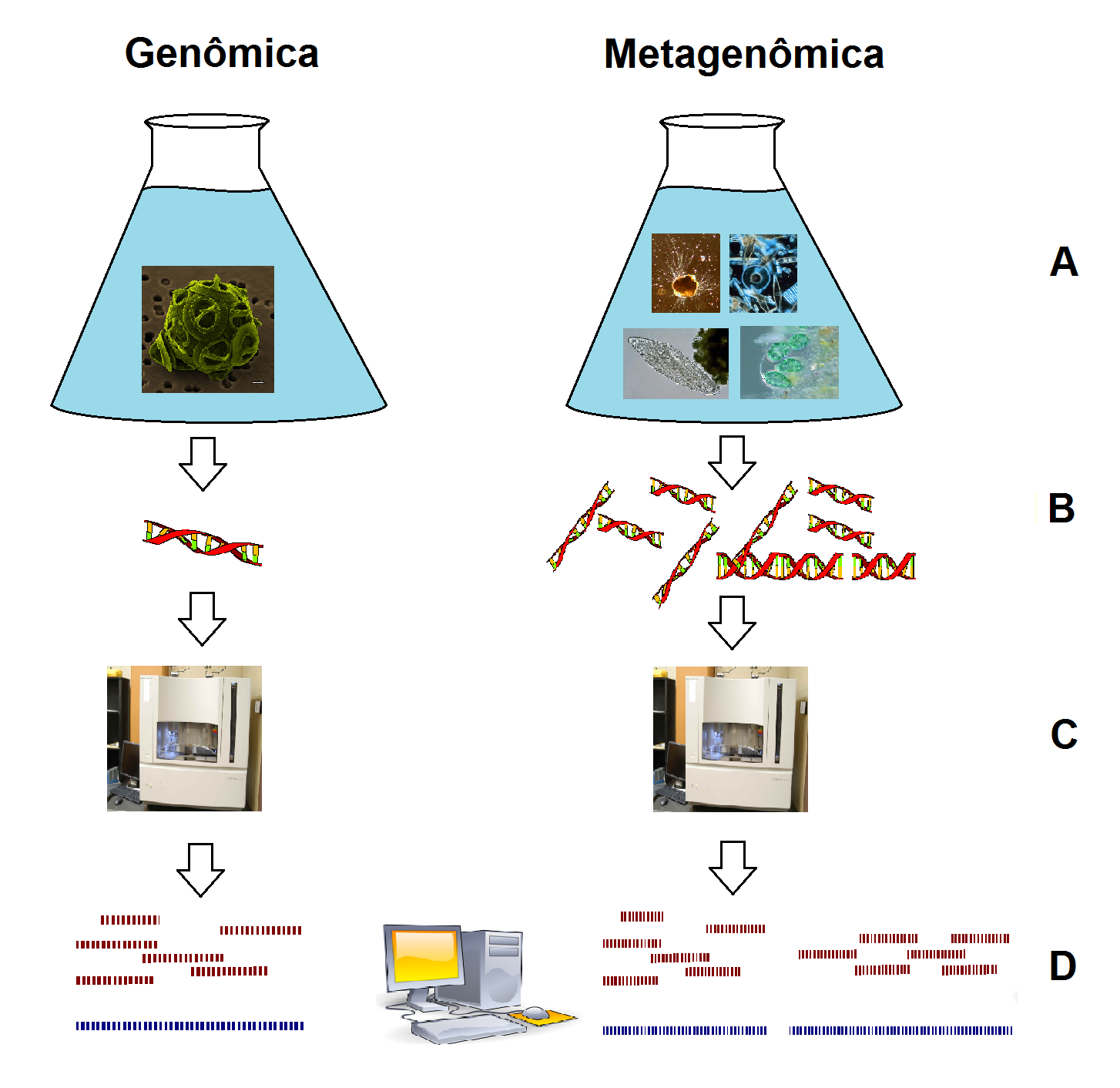 |
| Comparação entre a genômica e a metagenômica. Enquanto a primeira estuda o genoma de organismos (macro ou micro) individualmente, a metagenômica estuda o genoma de uma comunidade inteira de microorganismos, simultaneamente. (A) Amostras de microorganismos obtidos do ambiente. (B) Extração, cópia e fragmentação do DNA das amostras. (C) Sequenciamento dos fragmentos de DNA. (D) Montagem, num computador, das sequências dos fragmentos, obtendo-se as sequências das moléculas de DNA originais. Modificado de: http://commons.wikimedia.org/wiki/File:Gephyrocapsa_oceanica_color.jpg (CC BY-SA 2.5), http://commons.wikimedia.org/wiki/File:Paramecium_caudatum_Ehrenberg,_18... (CC BY-SA 3.0), http://commons.wikimedia.org/wiki/File:Glaucocystis_sp.jpg (CC BY-SA 2.5), http://commons.wikimedia.org/wiki/File:Ammonia_tepida.jpg (CC BY 2.5), http://commons.wikimedia.org/wiki/File:Diatoms_through_the_microscope.jpg (domínio public), |
Dentre os desafios para a metagenômica, está a busca de sequenciamentos ainda mais baratos e de tecnologias de bioinformática mais ágeis, o que facilitaria o estudo de microorganismos eucariotos (algas, protozoários, fungos). Atualmente, a maioria dos estudos foca nas bactérias, que são em geral pequenas e podem ser separadas dos eucariotos por filtração das amostras. Isto ocorre porque as bactérias possuem DNAs menores, ou seja, mais baratos de sequenciar e mais simples de se estudar.
(quadro) Saiba mais: Como o material genético vira letrinhas num computador?
Há vários métodos de sequenciamento de DNA, que diferem em custo e velocidade. Alguns são mais indicados para sequenciar pequenas sequências, outros para genomas inteiros. No método do pirossequenciamento, muito usado atualmente, primeiro isola-se o DNA dos outros componentes celulares. Faz-se várias cópias de cada macromolécula. Depois, quebra-se cada macromolécula de DNA em pedaços um pouco menores, aleatoriamente. Até aqui, as macromoléculas de DNA ainda possuem a estrutura de dupla fita (isto é, ainda são compostas, na verdade, por duas macromoléculas ou fitas alongadas). Então, a dupla fita é separada.
No próximo passo, os fragmentos de DNA, em fita simples, são transferidos para um recipiente em que há vários nucleotídeos (moléculas que são as subunidades do DNA) “colados” no fundo do recipiente. Em cada nucleotídeo “colado”, junta-se o nucleotídeo da ponta de um dos fragmentos de DNA. Então, adicionam-se mais nucleotídeos especiais (emitem luz quando pareados), que começarão a se parear com a porção livre de cada fragmento, reconstituindo a fita dupla. Cada nucleotídeo especial (A, T, C ou G) emite uma luz diferente quando se pareia, e seu sinal é detectado por um laser leitor.
Assim, o sinal é enviado para um computador, que vai anotando as sequências obtidas. Então, no computador, é feita a montagem das sequências dos fragmentos de DNA, obtendo-se a sequência das moléculas de DNA iniciais. Isto é possível graças às várias cópias de DNA feitas inicialmente, que geraram fragmentos redundantes, cujas sequências das extremidades se sobrepõem (são iguais) e permitem a sua montagem pelo computador.
As sequências dos genomas são então publicadas em bancos de dados online, de modo que outros pesquisadores possam usar os dados para suas pesquisas.